Liebe Eva,
danke für deinen Beitrag, der sicherlich für viele andere Nutzer hilfreich ist.
Mit pCourseClass in der SC2 hast du dir einen Datensatz ausgesucht, der tatsächlich ein paar Fallstricke bereithält und nicht ohne weiteres an andere Datensätze angespielt werden kann.
Im zugehörigen Datenmanual auf Seite 74f findest du hierzu schonmal ein paar Hinweise.
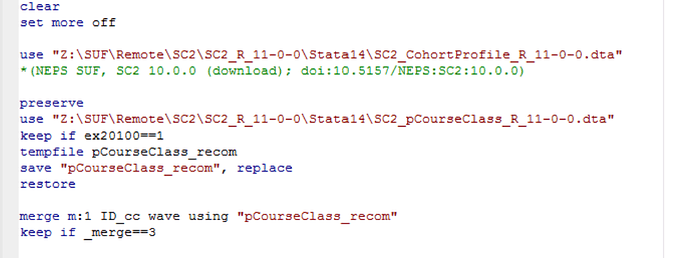
Zunächst gilt es, den Datensatz pCourseClass um Duplikate zu bereinigen. Dummerweise kam es im Feldverlauf dazu, dass teilweise manche Lehrer doppelte Fragebögen ausgefüllt haben oder mehrere Lehrer Fragebögen für dieselbe Klasse augefüllt haben.
use [Datenpfad]/pCourseClass.dta, clear
keep if ex20100 ==1
duplicates tag ID_cc wave, gen(dups) // doppelte Fälle markieren
bysort ID_cc dups: drop if dups ==1 & _n!=1 // so behält man nur jeweils die erste Angabe
tempfile class
save `class' // temporär abspeichern
Um diese Daten nun an CohortProfile mergen zu können, muss man zunächst sicherstellen, dass die beötigten merge-Variablen ID_cc und wave im Datensatz eindeutig die Beobachtungen identifizieren können. Da in CohortProfile allerdings auch Beobachtungen enthalten sind, in denen keine Klasseninfo erhoben wurde und die betreffende ID-Variable daher mit einem Missing für all diese Fälle befüllt ist, kann man nicht ohne Weiteres an CohortProfile anmergen.
Wir müssen daher alle Beobachtungen, die einen fehlenden Wert in ID_cc aufweisen, entfernen. Ob wir diese Fälle später wieder hinzufügen, hängt dann einfach von der geplanten Analyse ab. In der folgenden Syntax habe ich mal beide Optionen integriert:
use [Datenpfad]/CohortProfile.dta, clear
count if inlist(ID_cc,.,-55) // wie viele missings gibt es?
assert !inlist(ID_cc,.,-55) if tx80525 ==1 // wenn die Klasseninfo vorliegt, gibt es nur gültige Werte
// Option (1): Fälle ohne Klasseninfo sollen nach dem Merge wieder hinzugefügt werden können:
frame put _all if tx80525 ==0, into(fr_noclass) // in extra frame schieben
// ODER
// Option (2): alle Fälle ohne Klasseninfo direkt löschen
drop if tx80525 == 0
assert !inlist(ID_cc,.,-55) // sicherstellen, dass nur noch gültige Werte vorhanden sind
// da in CohortProfile mehrere Kinder derselben Klasse angehören und im Klassendatensatz
// nur je 1 Info pro Klasse vorliegt, benötigen wir einen m:1 merge!
merge m:1 ID_cc wave using `class' // vorher gespeicherten Datensatz anmergen
// optional: vorher gelöschte Fälle wieder hinzufügen:
frameappend fr_noclass
frame drop frnoclass
Alternativ könnte man auch einfach eine zusätzliche künstliche merge-Variable in pCourseClass anlegen, um direkt mit CohortProfile mergen zu können:
use [Datenpfad]/pCourseClass.dta, clear
keep if ex20100 ==1
duplicates tag ID_cc wave, gen(dups) // doppelte Fälle markieren
bysort ID_cc dups: drop if dups ==1 & _n!=1 // so behält man nur jeweils die erste Angabe
tempfile class
gen tx80525 = 1 // künstliche Identifikatorvariable erzeugen
save `class' // temporär abspeichern
use [Datenpfad]/CohortProfile.dta, clear
merge m:1 ID_cc wave tx80525 using `class'
Der Trick ist hier, dass man so tut als hätte man eine tx80525 Variable in beiden Datensätzen, weil man ja weiß, dass man nur an diejenigen Beobachtungen in CohortProfile mergen will, die tx80525 ==1 sind. Das ist vermutlich auch der kürzere und einfachere Weg. Sorry, dass mir der erst während des Schreibens eingefallen ist  Jetzt kannst du entscheiden, wie dus machen möchtest. Viele Wege führen zum Ziel!
Jetzt kannst du entscheiden, wie dus machen möchtest. Viele Wege führen zum Ziel!
Damit solltest du problemlos die Informationen des Klasslehrers mit analysieren können!
Viel Erfolg und beste Grüße,
Benno